اين سوالات، ابهامات و پيچيدگي ها، موجب شده است تا بعد از فراگير شدن استفاده از کامپيوتر و نرم افزارهاي کامپيوتري، زيست شناسان و تکامل شناسان از نرم افزارها و برنامه هاي کامپيوتري متعددي براي تلفيق و مرتبط کردن الگوريتم هاي ناهمگون بهره ببيند تا شايد بتوانند مشکلات ناشي از ناهماهنگي ها و تناقضات مشاهده شده در آناليز ژن هاي مختلف را به نحوي کاهش دهند!
بسم الله الرحمن الرحيم
ادامه ی مبحث فسيل هاي « نئاندرتال ها » و مطالعات ژنتيک
G) علاوه بر مطالبي که تاکنون پيرامون مشکلات مربوط به انواع مطالعات ژنتيکي انجام شده روي فسيل ها ذکر گرديد، مشکلات جدي نيز در مورد مطالعات مبتني بر پروتکل « DNA باستاني : Ancient DNA » استفاده کننده از DNA ميتوکندريال (mtDNA) وجود دارد.
DNA ميتوکندريال (mtDNA)، نقش مهمي در مطالعات مبتني بر روش « DNA باستاني : Ancient DNA » بازي مي کند و بسياري از آناليزهاي انجام شده بر روي DNA فسيل ها، با استفاده از DNA ميتوکندريال (mtDNA) صورت گرفته است و فسيل هاي « نئاندرتال ها » نيز از آن بي نصيب نبوده اند!(194)
DNA ميتوکندريال (mtDNA)، نقش مهمي در مطالعات مبتني بر روش « DNA باستاني : Ancient DNA » دارد و بسياري از آناليزهاي انجام شده بر روي DNA فسيل ها، با استفاده از DNA ميتوکندريال (mtDNA) صورت گرفته است و فسيل هاي « نئاندرتال ها » نيز از آن بي نصيب نبوده اند!
DNA ميتوکندريال (mtDNA)، نقش مهمي در مطالعات مبتني بر روش « DNA باستاني : Ancient DNA » دارد و بسياري از آناليزهاي انجام شده بر روي DNA فسيل ها، با استفاده از DNA ميتوکندريال (mtDNA) صورت گرفته است و فسيل هاي « نئاندرتال ها » نيز از آن بي نصيب نبوده اند!
استفاده از DNA ميتوکندريال (mtDNA) در مطالعات فسيلي مبتني بر روش « DNA باستاني : Ancient DNA »، به چند دليل مورد توجه بوده است: اولاً گرچه DNA ميتوکندريال (mtDNA)، درصد بسيار کمي از کل DNA سلولي را تشکيل مي دهد، اما همين درصد کم، در کپي هاي متعدد و فراوان در سطح سلول ها پخش شده اند(170) و بازيابي آن ها امکان پذير است. ثانياً به دليل اين که مطابق مفروضات علمي (تا سال 1995 ميلادي)، انتقال DNA ميتوکندريال (mtDNA)، از مادر به فرزندان در طي نسل ها رخ داده و « توارث مادري » دارد(173)، گزينه ي جذابي براي مطالعات تبارشناسي و از جمله بررسي زمان جدا شدن نسل ها از يکديگر به شمار مي رود.(173)
بنا بر مفروضات قبلي، DNA ميتوکندريال (mtDNA) عمدتاً تنها از مادران به فرزندان انتقال مي يابد و به همين ترتيب، عمدتاً تنها دختران اين مادران مي توانند DNA ميتوکندريال (mtDNA) کسب شده را به فرزندان خود انتقال دهند و … بدين ترتيب، عمدتاً تنها مادران و دختران مي توانند عامل انتقال نسل به نسل DNA ميتوکندريال به نسل بعد از خود باشند! به همين دليل توارث DNA ميتوکندريال (mtDNA) را « توارث مادري » مي نامند که مي تواند تنها از طريق مادران به دختران و سپس نواده هاي دختري و … انتقال يابد. با توجه به اين مفروضات، DNA ميتوکندريال (mtDNA) به عنوان يک گزينه ي جذاب براي و از جمله بررسي زمان جدا شدن نسل ها از يکديگر به شمار مي رود.
البته امروزه، کم کم مطالعه بر روي DNA هسته اي فسيل ها نيز رونق گرفته و اين نوع مطالعات در حال افزايش است!(195)
تا اين جاي کار براي مخاطبان محترم و به خصوص عزيزاني که مطالعاتي در حوزه ي « زيست شناسي » دارند، مسئله ي عجيب و غريبي نيست! اما موضوع هنگامي جالب مي شود که بدانيم، مفروضات قبلي در مورد DNA ميتوکندريال (mtDNA) از حوالي سال 1995 ميلادي به بعد، با ابهامات و ايرادات جدي موجه شده است که برخي از آن ها به شرح زير مي باشند:
الف) برخلاف تصورات قبلي در مورد اين که DNA ميتوکندريال (mtDNA) صرفاً به صورت توارث مادري انتقال مي يابد، شواهدي به دست آمده است که نشان مي دهد در برخي موارد، DNA ميتوکندريال (mtDNA) موجود در اسپرم پدر نيز بعد از لقاح مي تواند زنده مانده و به فرزندان انتقال يابد! (196)
برخلاف تصورات قبلي در مورد اين که DNA ميتوکندريال (mtDNA) صرفاً به صورت توارث مادري انتقال مي يابد، شواهدي به دست آمده است که نشان مي دهد در برخي موارد، DNA ميتوکندريال (mtDNA) موجود در اسپرم پدر نيز بعد از لقاح مي تواند زنده مانده و به فرزندان انتقال يابد!
برخلاف تصورات قبلي در مورد اين که DNA ميتوکندريال (mtDNA) صرفاً به صورت توارث مادري انتقال مي يابد، شواهدي به دست آمده است که نشان مي دهد در برخي موارد، DNA ميتوکندريال (mtDNA) موجود در اسپرم پدر نيز بعد از لقاح مي تواند زنده مانده و به فرزندان انتقال يابد!
بدين ترتيب، مفروضات قبلي در مورد اين که توارث DNA ميتوکندريال (mtDNA) صرفاً به صورت « توارث مادري » است، زير سوال مي رود و احتمال « توارث پدري » يا « توارث توام » نيز در اين ميان مطرح مي گردد!
ب) نکته ي ديگري که مفروضات قبلي را درباره ي DNA ميتوکندريال (mtDNA) زير سوال مي برد، کشف مسئله ي تنوع داخل سلولي ميتوکندري ها يا « هتروپلاسمي (ناهمگوني) : Heteroplasmy » است.
به عبارت ديگر، قبلاً تصور مي شد که تمامي ميتوکندري هاي داخل سلولي، کپي يکساني از DNA دارند؛ حال آن که در مطالعات بعدي مشخص گرديد که در بسياري از موجودات زنده و از جمله انسان ها، ممکن است ميتوکندري هاي مختلف داخل يک سلول، DNA هاي متفاوتي داشته باشند!:(197)
تصوير راست، نمايي از « هوموپلاسمي : Homoplasmy » را نشان مي دهد که در آن تمامي ميتوکندري ها (بيضي هاي زرد رنگ)، حاوي DNA يکساني هستند. قبلاً تصور مي شد که تمامي سلول هاي بدن موجودات زنده از جمله انسان، داراي وضعيت « هوموپلاسمي : Homoplasmy » هستند. تصاوير چپ، نمايي از « هتروپلاسمي (ناهمگوني) : Heteroplasmy » زا نشان مي دهند که در آن ها، ميتوکندري هاي سلول (بيضي هاي زرد رنگ وقرمز رنگ)، حاوي DNA هاي متفاوتي هستند! امروزه مشخص گرديده است که پديده ي « هتروپلاسمي (ناهمگوني) : Heteroplasmy »، يک پديده ي واقعي و شايع در سلول هاي بدن موجودات زنده و از جمله انسان است!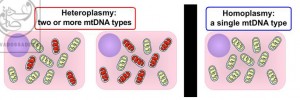
البته پديده ي « هتروپلاسمي (ناهمگوني) : Heteroplasmy » ميتوکندري ها، پديده ي ناشايعي نيست! بلکه برخي مطالعات، شيوع « هتروپلاسمي (ناهمگوني) : Heteroplasmy » ميتوکندري ها را در سلول هاي انسان ها، از 10 درصد الي 20 درصد افراد جوامع انساني گزارش نموده اند:(197)
پديده ي « هتروپلاسمي (ناهمگوني) : Heteroplasmy » ميتوکندري ها در داخل سلول ها، پديده ي ناشايعي نيست! بلکه برخي مطالعات، شيوع « هتروپلاسمي (ناهمگوني) : Heteroplasmy » ميتوکندري ها را در سلول هاي انسان ها، از 10 درصد الي 20 درصد گزارش نموده اند!
 اما نکته ي شگفت انگيز تر اين که حتي در بدن يک انسان نيز، ممکن است از بافتي به بافت ديگر، ميزان « هتروپلاسمي (ناهمگوني) : Heteroplasmy » ميتوکندري ها متغير باشد!(198) براي مثال، ممکن است ميزان « هتروپلاسمي (ناهمگوني) : Heteroplasmy » ميتوکندري ها در سلول هاي عضلات اسکلتي، با ميزان « هتروپلاسمي (ناهمگوني) : Heteroplasmy » ميتوکندري ها در سلول هاي پوست، متفاوت باشد! همچنين مشخص شده است که حتي با تغيير سن انسان نيز، ممکن است ميزان « هتروپلاسمي (ناهمگوني) : Heteroplasmy » ميتوکندري ها تغيير کند!:(198)
اما نکته ي شگفت انگيز تر اين که حتي در بدن يک انسان نيز، ممکن است از بافتي به بافت ديگر، ميزان « هتروپلاسمي (ناهمگوني) : Heteroplasmy » ميتوکندري ها متغير باشد!(198) براي مثال، ممکن است ميزان « هتروپلاسمي (ناهمگوني) : Heteroplasmy » ميتوکندري ها در سلول هاي عضلات اسکلتي، با ميزان « هتروپلاسمي (ناهمگوني) : Heteroplasmy » ميتوکندري ها در سلول هاي پوست، متفاوت باشد! همچنين مشخص شده است که حتي با تغيير سن انسان نيز، ممکن است ميزان « هتروپلاسمي (ناهمگوني) : Heteroplasmy » ميتوکندري ها تغيير کند!:(198)
 حتي در بدن يک انسان نيز، ممکن است از بافتي بهبافت ديگر، ميزان « هتروپلاسمي (ناهمگوني) : Heteroplasmy » ميتوکندري ها متغير باشد! براي مثال، ممکن است ميزان « هتروپلاسمي (ناهمگوني) : Heteroplasmy » ميتوکندري هادر سلول هاي عضلات اسکلتي، با ميزان « هتروپلاسمي (ناهمگوني) : Heteroplasmy » ميتوکندري ها در سلول هاي پوست، متفاوت باشد! همچنين مشخص شده است که حتي با تغيير سن انسان نيز، ممکن است ميزان « هتروپلاسمي (ناهمگوني) : Heteroplasmy » ميتوکندري ها تغيير کند!
حتي در بدن يک انسان نيز، ممکن است از بافتي بهبافت ديگر، ميزان « هتروپلاسمي (ناهمگوني) : Heteroplasmy » ميتوکندري ها متغير باشد! براي مثال، ممکن است ميزان « هتروپلاسمي (ناهمگوني) : Heteroplasmy » ميتوکندري هادر سلول هاي عضلات اسکلتي، با ميزان « هتروپلاسمي (ناهمگوني) : Heteroplasmy » ميتوکندري ها در سلول هاي پوست، متفاوت باشد! همچنين مشخص شده است که حتي با تغيير سن انسان نيز، ممکن است ميزان « هتروپلاسمي (ناهمگوني) : Heteroplasmy » ميتوکندري ها تغيير کند!
بدين ترتيب با توجه به مطالعات انجام شده در طي سال هاي اخير، مشخص گرديده است که مفروضات قبلي تکامل شناسان، در رابطه با « توارث DNA يکسان مادري » از مادر به فرزندان، نادرست بوده و موارد متعدد و اثبات شده اي از « هتروپلاسمي (ناهمگوني) : Heteroplasmy » ميتوکندري ها در طي سال هاي اخير کشف گرديده است! به نحوي که نه تنها در افراد يک خانواده، بلکه حتي در سلول هاي مختلف يک انسان و حتي در سنين مختلف عمر وي، ميزان متغير و گهگاه غيرقابل پيش بيني از « هتروپلاسمي (ناهمگوني) : Heteroplasmy » ميتوکندري ها ملاحظه مي گردد!!! با عنايت به اين کشفيات، مي توان گفت که مفروضات قبلي تکامل شناسان پيرامون « توارث مادري DNA ميتوکندريال (mtDNA) »، با چالش ها و اشکالات جدي مواجه است!
ج) مسئله ي ديگري که در مقابل ادعاهاي تکامل شناسان، چالش جدي ايجاد مي نمايد، اين نکته است که برخلاف مفروضات قبلي تکامل شناسان که سرعت بروز جهش در DNA ميتوکندريال (mtDNA) را طي نسل هاي مختلف، ثابت و يکسان فرض مي کردند، امروزه مشخص شده است که سرعت جهش هاي ژنتيکي در « DNA ميتوکندريال (mtDNA) »، متغير و غير قابل پيش بيني بوده و در برخي از موارد، سرعت بروز جهش ها در « DNA ميتوکندريال (mtDNA) »، بسيار بيشتر از آن چيزي است که در محاسبات قبلي تکامل شناسان، لحاظ شده بود!(199)
در واقع مطالعات جديد (پس از سال 1995 ميلادي) نشان داده اند که جهش هاي ژنتيکي يا « موتاسيون : Mutation » ها(172) در « DNA ميتوکندريال (mtDNA) »، بسيار سريع تر از محاسبات قبلي، به وقوع مي پيوندند!:(199)
مسئله ي ديگري که در مقابل ادعاهاي تکامل شناسان، چالش جدي ايجاد مي نمايد، اين نکته است که برخلاف مفروضات قبلي تکامل شناسان که سرعت بروز جهش در DNA ميتوکندريال (mtDNA) را طي نسل هاي مختلف، ثابت و يکسان فرض مي کردند، امروزه مشخص شده است که سرعت جهش هاي ژنتيکي در « DNA ميتوکندريال (mtDNA) »، متغير و غير قابل پيش بيني بوده و در برخي از موارد، سرعت بروز جهش ها در « DNA ميتوکندريال (mtDNA) »، بسيار بيشتر از آن چيزي است که در محاسبات تکامل شناسان لحاظ شده بود! در واقع مطالعات جديد (پس از سال 1995 ميلادي) نشان داده اند که جهش هاي ژنتيکي يا « موتاسيون : Mutation » ها در « DNA ميتوکندريال (mtDNA) »، بسيار سريع تر از محاسبات قبلي، به وقوع مي پيوندند!
مسئله ي ديگري که در مقابل ادعاهاي تکامل شناسان، چالش جدي ايجاد مي نمايد، اين نکته است که برخلاف مفروضات قبلي تکامل شناسان که سرعت بروز جهش در DNA ميتوکندريال (mtDNA) را طي نسل هاي مختلف، ثابت و يکسان فرض مي کردند، امروزه مشخص شده است که سرعت جهش هاي ژنتيکي در « DNA ميتوکندريال (mtDNA) »، متغير و غير قابل پيش بيني بوده و در برخي از موارد، سرعت بروز جهش ها در « DNA ميتوکندريال (mtDNA) »، بسيار بيشتر از آن چيزي است که در محاسبات تکامل شناسان لحاظ شده بود! در واقع مطالعات جديد (پس از سال 1995 ميلادي) نشان داده اند که جهش هاي ژنتيکي يا « موتاسيون : Mutation » ها در « DNA ميتوکندريال (mtDNA) »، بسيار سريع تر از محاسبات قبلي، به وقوع مي پيوندند!
همان گونه که ملاحظه فرموديد، با توجه به کشفيات جديد علم ژنتيک، سرعت بروز جهش هاي ژنتيکي در « DNA ميتوکندريال (mtDNA) » طي نسل هاي مختلف، متغير بوده و عموماً جهش ها سريع تر از آن چيزي که قبلاً تصور مي شد، به وقوع مي پيوندند!(199) اين کشفيات، ادعاهاي تکامل شناسان را که بر اساس مفروضات غلط قبلي شکل گرفته است، باطل مي کند و محاسبات صورت گرفته توسط آنان را که بر اساس پيش فرض هاي قبلي انجام شده است، مبدل به کاغذپاره مي نمايد!
اما از مجموع مطالبي که در بخش هاي (الف)، (ب) و (ج) گفته شد، اين نتيجه به دست مي آيد که ادعاهاي تکامل شناسان در حوزه هاي تبارشناسي و تکامل، کاملاً سست و ضعيف بوده و بر اساس اطلاعات قبل از سال 1995 ميلادي مي باشد! (گر چه هنوز تکامل شناسان با بي شرمي کامل از آن ها در کتب و مقالاتشان استفاده مي کنند و حتي اشاره اي به کشفيات جديد در اين حوزه نمي نمايند!).
امروزه بسياري از دانشمندان، اين حقيقت را پذيرفته اند که مطالعه در حوزه ي « DNA ميتوکندريال (mtDNA) »، بسيار پيچيده تر از آن است که قبلاً تصور مي شد؛(200) همچنين اين محققان معتقدند که ادعاهاي ساده انگارانه ي قبلي مي بايست، تعديل گردد و راهکارهاي نويني براي مطالعات جديد، در خدمت گرفته شود.(200)
از سوي ديگر، تعدادي از محققين نيز به صراحت درباره ي عواقبي که اين کشفيات جديد براي « تکامل شناسان » در پي دارد، اشاره کرده اند و اين کشفيات را موجب نگراني « تکامل شناسان » دانسته اند!:(200)
امروزه بسياري از دانشمندان، اين حقيقت را پذيرفته اند که مطالعه در حوزه ي « DNA ميتوکندريال (mtDNA) »، بسيار پيچيده تر از آن است که قبلاً تصور مي شد؛ همچنين اين محققان معتقدند که ادعاهاي ساده انگارانه ي قبلي مي بايست، تعديل گردد و راهکارهاي نويني براي مطالعات جديد، در خدمت گرفته شود. از سوي ديگر، تعدادي از محققين نيز به صراحت درباره ي عواقبي که اين کشفيات جديد براي « تکامل شناسان » در پي دارد، اشاره کرده اند و اين کشفيات را موجب نگراني « تکامل شناسان » دانسته اند!
امروزه بسياري از دانشمندان، اين حقيقت را پذيرفته اند که مطالعه در حوزه ي « DNA ميتوکندريال (mtDNA) »، بسيار پيچيده تر از آن است که قبلاً تصور مي شد؛ همچنين اين محققان معتقدند که ادعاهاي ساده انگارانه ي قبلي مي بايست، تعديل گردد و راهکارهاي نويني براي مطالعات جديد، در خدمت گرفته شود. از سوي ديگر، تعدادي از محققين نيز به صراحت درباره ي عواقبي که اين کشفيات جديد براي « تکامل شناسان » در پي دارد، اشاره کرده اند و اين کشفيات را موجب نگراني « تکامل شناسان » دانسته اند!
امروزه بسياري از دانشمندان، اين حقيقت را پذيرفته اند که مطالعه در حوزه ي « DNA ميتوکندريال (mtDNA) »، بسيار پيچيده تر از آن است که قبلاً تصور مي شد؛ همچنين اين محققان معتقدند که ادعاهاي ساده انگارانه ي قبلي مي بايست، تعديل گردد و راهکارهاي نويني براي مطالعات جديد، در خدمت گرفته شود. از سوي ديگر، تعدادي از محققين نيز به صراحت درباره ي عواقبي که اين کشفيات جديد براي « تکامل شناسان » در پي دارد، اشاره کرده اند و اين کشفيات را موجب نگراني « تکامل شناسان » دانسته اند!
امروزه بسياري از دانشمندان، اين حقيقت را پذيرفته اند که مطالعه در حوزه ي « DNA ميتوکندريال (mtDNA) »، بسيار پيچيده تر از آن است که قبلاً تصور مي شد؛ همچنين اين محققان معتقدند که ادعاهاي ساده انگارانه ي قبلي مي بايست، تعديل گردد و راهکارهاي نويني براي مطالعات جديد، در خدمت گرفته شود. از سوي ديگر، تعدادي از محققين نيز به صراحت درباره ي عواقبي که اين کشفيات جديد براي « تکامل شناسان » در پي دارد، اشاره کرده اند و اين کشفيات را موجب نگراني « تکامل شناسان » دانسته اند!
بدين ترتيب همان گونه که ملاحظه فرموديد، ادعاهاي تکامل شناسان پيرامون دقت بالاي مطالعات مبتني بر « DNA ميتوکندريال (mtDNA) »، با ابهامات و چالش هاي جدي مواجه شده و با توجه به اين که 50 درصد يا حتي بيش از 50 درصد مطالعات مبتني بر روش « DNA باستاني : Ancient DNA » که تاکنون انجام شده اند، بر اساس « DNA ميتوکندريال (mtDNA) » و با توجه به مفروضات قبلي، طرح ريزي شده اند، بايد گفت که حداقل نيمي از مطالعات ژنتيکي مبتني بر روش « DNA باستاني : Ancient DNA » که تاکنون انجام شده اند، با چالش ها، ابهامات و سوالات جدي مواجه هستند!
گرچه برخي مشکلات ذکر شده در مورد « DNA ميتوکندريال (mtDNA) » همچون « هتروپلاسمي » در مورد DNA هسته اي وجود ندارد، اما ساير مشکلاتي که قبلاً در مورد مشکلات موجود در روش « DNA باستاني : Ancient DNA » ذکر شد – اعم از تغييرات شيميايي DNA بعد از مرگ، آلودگي نمونه هاي DNA، استاندارد نبودن روش هاي مطالعه بر روي DNA باستاني، تعداد کم فسيل هاي مورد مطالعه، و ورود DNA ويروس به DNA بدن انسان و جانوران (که اتفاقاً اختصاص به DNA هسته اي دارد) -، هم در DNA ميتوکندريال (mtDNA) و هم در DNA هسته اي به چشم مي خورد و مطالعات مربوط به هر دو نوع DNA را به چالش مي طلبد! بدين ترتيب هر دو نوع DNA در مطالعات مبتني بر روش « DNA باستاني : Ancient DNA » با مشکلات جدي رو به رو هستند!
H) اما يکي ديگر از مشکلاتي که در مورد مطالعات مبتني بر روش « DNA باستاني : Ancient DNA » وجود دارد، مسئله ي عدم تطابق يافته هاي حاصل از مطالعه بر روي ژن ها و قطعات مختلف DNA است! در واقع اين مشکل که از يک سو، هم در مطالعات « DNA باستاني : Ancient DNA » و هم در مطالعات DNA موجودات امروزي وجود دارد و از سوي ديگر، هم مطالعات انجام شده بر روي DNA ميتوکندريال (mtDNA) و هم مطالعات مربوط به DNA هسته اي را با چالش مواجه مي کند، موجب بروز اشکالات، ابهامات و سردرگمي هاي بسيار مي گردد!
قبل از بررسي اين مشکل، مي بايست توضيح مختصري پيرامون « درخت فيلوژنتيک : Phylogenetic tree »(201)، « درخت اتصال – همسايگي : tree Neighbor joining »(202) و « درخت بايزي : Bayesian tree »(203) بدهيم؛ « درخت فيلوژنتيک : Phylogenetic tree »(201)، « درخت اتصال – همسايگي : Neighbor joining »(202) و « درخت بايزي : Bayesian tree »(203)، يکي از شايع ترين متدها و روش هايي هستند که « تکامل شناسان » از آن ها براي نشان دادن رابطه ي خويشاوندي موجودات بهره مي برند. بدين ترتيب که موجودات زنده ي مد نظر خود را در « درخت » هايي به نمايش مي گذارند که هر شاخه ي اين درخت ها، متعلق به يک « موجود زنده » مي باشد. در اين درخت ها، هر چه موجودات از نظر تکاملي به هم نزديکتر باشند و هر چه خويشاوندي بيشتر و شباهت ظاهري (آناتوميک)، فيزيولوژيک يا ژنتيک بيشتري داشته باشند، در شاخه هاي نزديک تري قرار مي گيرند و هر چه از نظر تکاملي از يکديگر دورتر باشند و هر چه خويشاوندي کمتر، شباهت ظاهري (آناتوميک)، فيزيولوژيک يا ژنتيک کمتري داشته و از همديگر دورتر باشند، در شاخه هاي دورتري نسبت به يکديگر قرار مي گيرند (البته به زعم تکامل شناسان!):
تصاويري از « درخت فيلوژنتيک : Phylogenetic tree »، « درخت اتصال – همسايگي : Neighbor joining tree » و « درخت بايزي : Bayesian tree »؛ هر شاخه ي اين درخت ها، متعلق به يک « موجود زنده » مي باشد. در اين درخت ها، هر چه موجودات از نظر تکاملي به هم نزديکتر باشند و هر چه خويشاوندي بيشتر و شباهت ظاهري (آناتوميک)، فيزيولوژيک يا ژنتيک بيشتري داشته باشند، در شاخه هاي نزديک تري قرار مي گيرند (موجودات زنده ي داخل کادر قرمز رنگ) و هر چه از نظر تکاملي از يکديگر دورتر باشند و هر چه خويشاوندي کمتر، شباهت ظاهري (آناتوميک)، فيزيولوژيک يا ژنتيک کمتري داشته و از همديگر دورتر باشند – به زعم تکامل شناسان! -، در شاخه هاي دورتري نسبت به يکديگر قرار مي گيرند (فاصله ي بين موجودات زنده ي کادر قرمز رنگ و کادر آبي رنگ)
البته از نظر علمي، اختلافاتي بين تعاريف « درخت فيلوژنتيک : Phylogenetic tree »(201)، « درخت اتصال – همسايگي : Neighbor joining tree »(202) و « درخت بايزي : Bayesian tree »(203) وجود دارد که از ذکر آن به دليل جلوگيري از طولاني شدن بحث خودداري مي نماييم؛ اما معمولاً « درخت فيلوژنتيک : Phylogenetic tree »(201) براي مقاصد مقايسه ي ظاهري (آناتوميک) و ژنتيک کاربرد دارد، اما « درخت اتصال – همسايگي : Neighbor joining tree »(202) و « درخت بايزي : Bayesian tree »(203) بيشتر براي مقاصد مقايسه ي ژنتيکي يا ساختار پروتئيني و امثالهم کاربرد دارند.
به هر حال، آن چه اهميت دارد، اين است که نزديکي يا دوري ظاهري (آناتوميک)، ژنتيکي، پروتئوميکي و … موجودات زنده (البته به زعم تکامل شناسان!) به وسيله ي درخت هايي همچون « درخت فيلوژنتيک : Phylogenetic tree »(201)، « درخت اتصال – همسايگي : Neighbor joining tree »(202) و « درخت بايزي : Bayesian tree »(203) نمايش داده مي شود که در اين درختان، دوري يا نزديکي شاخه ها ، معرف دوري يا نزديکي موجودات زنده ي مورد نظر است.
قبلاً به مشکل عدم شباهت تقسيم بندي و درخت فيلوژنتيکي اسب هاي موسوم به اسب هاي ماقبل تاريخ بر اساس ظاهر فسيل هاي آن ها، با تقسيم بندي و درخت فيلوژنتيکي آن ها بر اساس آناليز به روش « DNA باستاني : Ancient DNA » در بخش هاي قبلي همين مقاله اشاره کرديم.
اما مشکل هنگامي بيشتر خود را نشان مي دهد که ملاحظه مي گردد که مطالعات مختلف انجام شده بر روي بخش هاي مختلفي از DNA و بخش هاي مختلفي از ژن هاي گوناگون فسيل ها يا موجودات زنده ي کنوني، نتايج متفاوت و متغيري ارايه مي دهند! براي مثال هنگامي که 3 موجود زنده ي A، B و C از نظر ژنتيکي بررسي مي گردند، نتايج آناليز بر روي ژن (الف) اين موجودات زنده، با نتايج آناليز بر روي ژن (ب) اين موجودات زنده، متفاوت مي باشد!
تصاوير زير، « درخت هاي فيلوژنتيک : Phylogenetic trees » چند گونه از « پلاسموديوم : Plasmodium » ها را که انواع انساني آن در انسان، بيماري « مالاريا » را ايجاد مي کند، بر اساس 3 پارامتر شامل « DNA هسته اي کد کننده ي زير واحد کوچک RNA ريبوزومي (rRNA) موسوم به (SSU) »، « DNA ميتوکندريال (mtDNA) کد کننده ي پروتئين سيتوکروم b موسوم به (Cytochrome b) » و نيز « DNA پلاستيدي کد کننده ي پروتئاز کازئينوليتيک موسوم به (ClpC) » نشان مي دهد:(204)
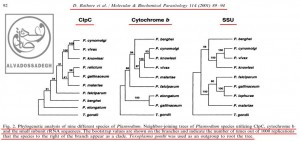 تصاوير فوق، « درخت هاي فيلوژنتيک : Phylogenetic trees » چند گونه از « پلاسموديوم : Plasmodium » ها را که انواع انساني آن در انسان بيماري « مالاريا » را ايجاد مي کند، بر اساس 3 پارامتر شامل « DNA هسته اي کد کننده ي زير واحد کوچک RNA ريبوزومي (rRNA) موسوم به (SSU) »، « DNA ميتوکندريال (mtDNA) کد کننده ي پروتئين سيتوکروم b موسوم به (Cytochrome b) » و نيز « DNA پلاستيدي کد کننده ي پروتئاز کازئينوليتيک موسوم به (ClpC) » نشان مي دهد.
تصاوير فوق، « درخت هاي فيلوژنتيک : Phylogenetic trees » چند گونه از « پلاسموديوم : Plasmodium » ها را که انواع انساني آن در انسان بيماري « مالاريا » را ايجاد مي کند، بر اساس 3 پارامتر شامل « DNA هسته اي کد کننده ي زير واحد کوچک RNA ريبوزومي (rRNA) موسوم به (SSU) »، « DNA ميتوکندريال (mtDNA) کد کننده ي پروتئين سيتوکروم b موسوم به (Cytochrome b) » و نيز « DNA پلاستيدي کد کننده ي پروتئاز کازئينوليتيک موسوم به (ClpC) » نشان مي دهد.
همان گونه که در تصاوير فوق مشاهده مي فرماييد، علي رغم اين که طبق ادعاهاي تکامل شناسان، انتظار مي رود که در مقايسه بين « گونه » هاي مختلف انگل « پلاسموديوم »، آناليز بخش هاي مختلف ژني و DNAهاي قسمت هاي مختلف سلول هاي آن ها (اعم از DNA هسته اي، DNA ميتوکندريال (mtDNA) و DNA پلاستيدي)، درخت هاي فيلوژنتيک کم و بيش مشابهي به دست آيد – تا همگام با هم سير خويشاوندي و تکاملي مشابهي به نمايش بگذارند -، اما ملاحظه مي گردد که آناليز هر DNA، درخت فيلوژنتيک متفاوتي را به نمايش مي گذارد!!!
براي مثال، در آناليز به دست آمده از « DNA ميتوکندريال (mtDNA) کد کننده ي پروتئين سيتوکروم b موسوم به (Cytochrome b) »، مشاهده مي شود که دو گونه ي « پلاسموديوم الونگاتوم : P. elongatum » و « پلاسموديوم گاليناسئوم : P. gallinaceum » قرابت زيادي با همديگر از نظر اين ژن دارند و بر روي دو شاخه ي مجاور قرار دارند، اما همين دو گونه يعني « پلاسموديوم الونگاتوم : P. elongatum » و « پلاسموديوم گاليناسئوم : P. gallinaceum » هنگامي که از نظر « DNA پلاستيدي کد کننده ي پروتئاز کازئينوليتيک موسوم به (ClpC) » بررسي مي گردند، تفاوت هاي فاحشي نشان مي دهند و بر روي دو شاخه ي دور از هم قرار مي گيرند!(204)
مثال ديگر در همين آناليز ها، مقايسه ي بين دو گونه ي « پلاسموديوم گاليناسئوم : P. gallinaceum » و « پلاسموديوم مالاريه : P. malariae » مي باشد؛ به نحوي که آناليز « DNA پلاستيدي کد کننده ي پروتئاز کازئينوليتيک موسوم به (ClpC) »، جايگاه « پلاسموديوم گاليناسئوم : P. gallinaceum » و « پلاسموديوم مالاريه : P. malariae » را در شاخه هاي به نسبت نزديک به هم و داراي قرابت زياد نشان مي دهد، اما آناليز « DNA هسته اي کد کننده ي زير واحد کوچک RNA ريبوزومي (rRNA) موسوم به (SSU) » و « DNA ميتوکندريال (mtDNA) کد کننده ي پروتئين سيتوکروم b موسوم به (Cytochrome b) » براي همين دو گونه، جايگاه به مراتب دورتري را به نمايش مي گذارد:(204)
همان گونه که در تصاوير فوق مشاهده مي فرماييد، علي رغم اين که با توجه به ادعاهاي تکامل شناسان، انتظار مي رود که در مقايسه بين « گونه » هاي مختلف انگل « پلاسموديوم »، آناليز بخش هاي مختلف ژني و DNAهاي قسمت هاي مختلف سلول هاي آن ها (اعم از DNA هسته اي، DNA ميتوکندريال (mtDNA) و DNA پلاستيدي)، درخت هاي فيلوژنتيک کم و بيش مشابهي به دست آيد – تا همگام با هم سير خويشاوندي و تکاملي مشابهي به نمايش بگذارند -، اما ملاحظه مي گردد که آناليز هر DNA، درخت فيلوژنتيک متفاوتي را به نمايش مي گذارد!!! در آناليز به دست آمده از « DNA ميتوکندريال (mtDNA) کد کننده ي پروتئين سيتوکروم b موسوم به (Cytochrome b) »، مشاهده مي شود که دو گونه ي « پلاسموديوم الونگاتوم : P. elongatum » (کادر قرمز رنگ) و « پلاسموديوم گاليناسئوم : P. gallinaceum » (کادر سبز رنگ) قرابت زيادي با همديگر از نظر اين ژن دارند و بر روي دو شاخه ي مجاور قرار دارند، اما همين دو گونه يعني « پلاسموديوم الونگاتوم : P. elongatum » و « پلاسموديوم گاليناسئوم : P. gallinaceum » هنگامي که از نظر « DNA پلاستيدي کد کننده ي پروتئاز کازئينوليتيک موسوم به (ClpC) » بررسي مي گردند، تفاوت هاي فاحشي نشان مي دهند و بر روي دو شاخه ي دور از هم قرار مي گيرند! مثال ديگر در همين آناليز ها مقايسه ي بين دو گونه ي « پلاسموديوم گاليناسئوم : P. gallinaceum » (کادر سبز رنگ) و « پلاسموديوم مالاريه : P. malariae » (کادر آبي رنگ) مي باشد؛ به نحوي که آناليز « DNA پلاستيدي کد کننده ي پروتئاز کازئينوليتيک موسوم به (ClpC) »، جايگاه « پلاسموديوم گاليناسئوم : P. gallinaceum » و « پلاسموديوم مالاريه : P. malariae » را در شاخه هاي به نسبت نزديک به هم و داراي قرابت زياد نشان مي دهد، اما آناليز « DNA هسته اي کد کننده ي زير واحد کوچک RNA ريبوزومي (rRNA) موسوم به (SSU) » و « DNA ميتوکندريال (mtDNA) کد کننده ي پروتئين سيتوکروم b موسوم به (Cytochrome b) » براي همين دو گونه، جايگاه به مراتب دورتري را به نمايش مي گذارد!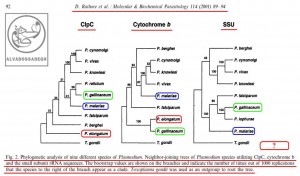
البته با دقت بيشتر در درخت هاي فيلوژنتيک ترسيم شده براي گونه هاي مختلف « پلاسموديوم » ها، باز هم موارد زيادي از اين گونه تناقضات و ناهماهنگي ها، مشخص مي گردد! اين مسئله نشان مي دهد که بسته به اين که کدام DNA سلولي مورد مطالعه واقع شود و حتي بسته به اين که کدام قطعه از DNA يا کدام ژن بررسي گردد، درخت هاي فيلوژنتيک رسم شده، مي تواند متفاوت باشد! و اين واقعيتي است که نه تنها براي درخت هاي فيلوژنتيک مربوط به « پلاسموديوم ها »، بلکه براي ژن هاي متفاوت موجودات زنده ي ديگر اعم از دلفين، خفاش، اسفنج، انسان و … نيز به چشم مي خورد که ان شاء الله در مقالات آتي که مفصلاً به بحث « ژنتيک و تکامل » مربوط مي شود، به مواردي از آن ها اشاره خواهيم کرد!
بدين ترتيب مشخص مي گردد که در بررسي هاي ژنتيکي و مطالعات مبتني بر DNA، درخت هاي فيلوژنتيک مربوط به بررسي هاي خويشاوندي و تکاملي، همواره نتايج يکسان و حتي مشابهي به دست نمي دهند! بلکه بستگي به اين که کدام قطعه از DNA يا کدام ژن موجودات زنده، با يکديگر مقايسه شود، نتايج متفاوت و درخت هاي فيلوژنتيک گوناگوني حاصل مي گردد که اين امر با ادعاهاي تکامل شناسان، قرابت زيادي ندارد! چرا که اگر سخنان آن ها در مورد قرابت برخي از موجودات زنده با يکديگر و دوري برخي از آن ها از يکديگر درست باشد، اصولاً نبايد تفاوت هاي زيادي در بررسي هاي ژنتيکي ژن هاي مختلف آن ها وجود داشته باشد! حال آن که در بسياري از موارد، درخت هاي فيلوژنتيک رسم شده بر اساس ژن هاي مختلف چند موجود زنده ي « به اصطلاح هم خانواده »، تفاوت هاي فاحشي را به نمايش مي گذارند!
اين سخن تنها ادعايي از جانب ما نيست؛ بلکه مقالات و مطالعات متعدد علمي نيز به وجود چنين ناهماهنگي هايي به صراحت اشاره کرده و در صدد اختراع و ابداع روش هاي آناليز، نرم افزار هاي کامپيوتري و ترفندهاي آماري هستند تا بتوانند مشکلات ناشي از اين ناهماهنگي ها را به حداقل برسانند!:(205)
در بررسي هاي ژنتيکي و مطالعات مبتني بر DNA، درخت هاي فيلوژنتيک مربوط به بررسي هاي خويشاوندي و تکاملي، همواره نتايج يکسان و حتي مشابهي به دست نمي دهند! بلکه بستگي به اين که کدام قطعه از DNA يا کدام ژن موجودات زنده، با يکديگر مقايسه شود، نتايج متفاوت و درخت هاي فيلوژنتيک گوناگوني حاصل مي گردد که اين امر با ادعاهاي تکامل شناسان، قرابت زيادي ندارد! چرا که اگر سخنان آن ها در مورد قرابت برخي از موجودات زنده با يکديگر و دوري برخي از آن ها از يکديگر درست باشد، اصولاً نبايد تفاوت هاي زيادي در بررسي هاي ژنتيکي ژن هاي مختلف آن ها وجود داشته باشد! حال آن که در بسياري از موارد، درخت هاي فيلوژنتيک رسم شده بر اساس ژن هاي مختلف چند موجود زنده ي « به اصطلاح هم خانواده »، تفاوت هاي فاحشي را به نمايش مي گذارند! اين سخن تنها ادعايي از جانب ما نيست؛ بلکه مقالات و مطالعات متعدد علمي نيز به وجود چنين ناهماهنگي هايي به صراحت اشاره کرده و در صدد اختراع و ابداع روش هاي آناليز، نرم افزار هاي کامپيوتري و ترفندهاي آماري هستند تا بتوانند مشکلات ناشي از اين ناهماهنگي ها را به حداقل برسانند!
در بررسي هاي ژنتيکي و مطالعات مبتني بر DNA، درخت هاي فيلوژنتيک مربوط به بررسي هاي خويشاوندي و تکاملي، همواره نتايج يکسان و حتي مشابهي به دست نمي دهند! بلکه بستگي به اين که کدام قطعه از DNA يا کدام ژن موجودات زنده، با يکديگر مقايسه شود، نتايج متفاوت و درخت هاي فيلوژنتيک گوناگوني حاصل مي گردد که اين امر با ادعاهاي تکامل شناسان، قرابت زيادي ندارد! چرا که اگر سخنان آن ها در مورد قرابت برخي از موجودات زنده با يکديگر و دوري برخي از آن ها از يکديگر درست باشد، اصولاً نبايد تفاوت هاي زيادي در بررسي هاي ژنتيکي ژن هاي مختلف آن ها وجود داشته باشد! حال آن که در بسياري از موارد، درخت هاي فيلوژنتيک رسم شده بر اساس ژن هاي مختلف چند موجود زنده ي « به اصطلاح هم خانواده »، تفاوت هاي فاحشي را به نمايش مي گذارند! اين سخن تنها ادعايي از جانب ما نيست؛ بلکه مقالات و مطالعات متعدد علمي نيز به وجود چنين ناهماهنگي هايي به صراحت اشاره کرده و در صدد اختراع و ابداع روش هاي آناليز، نرم افزار هاي کامپيوتري و ترفندهاي آماري هستند تا بتوانند مشکلات ناشي از اين ناهماهنگي ها را به حداقل برسانند!
بدين ترتيب همان گونه که ملاحظه فرموديد، مطالعات ژنتيکي بر روي ژن هاي مختلف موجودات زنده، نه تنها نتوانسته معماي خويشاوندي و ارتباط تکاملي موجودات زنده را حل کند، بلکه سوالات، ابهامات و پيچيدگي هاي زيادي را به همراه داشته است!
اين سوالات، ابهامات و پيچيدگي ها، موجب شده است تا بعد از فراگير شدن استفاده از کامپيوتر و نرم افزارهاي کامپيوتري، زيست شناسان و تکامل شناسان از نرم افزارها و برنامه هاي کامپيوتري متعددي براي تلفيق و مرتبط کردن الگوريتم هاي ناهمگون بهره ببيند تا شايد بتوانند مشکلات ناشي از ناهماهنگي ها و تناقضات مشاهده شده در آناليز ژن هاي مختلف را به نحوي کاهش دهند!(205) اما هنوز برنامه اي که بتواند بر اين ناهمگوني ها غلبه کند، ابداع نشده است!(205) همچنين برخي از محققين با مشاهده ي ناتواني الگوريتم هاي مختلف در متحد ساختن اطلاعات حاصل از آناليز هاي ژنتيکي، عنوان کرده اند که اصولاً ممکن است نتوان داده هاي حاصل از آناليزهاي ژنتيکي را در درخت هاي فيلوژنتيک که کامل ترين آن ها به عنوان « درخت فيلوژنتيک زندگي : Phylogenetic Tree of Life » ناميده مي شود، قرار داد!(205)
البته چنين نتايجي کاملاً قابل انتظار است! اصولاً تا زماني که تمامي موجودات زنده ي کره ي زمين کشف نشوند، نمي توان الگوريتم مناسبي که الگوهاي ژنتيکي موجودات زنده ي مختلف را پوشش دهد، طراحي نمود!
به هر حال ناسازگاري هاي موجود بين نتايج حاصل از مطالعات ژنتيکي و درخت هاي فيلوژنتيک مبتني بر DNA موجودات زنده، چالشي بزرگ براي زيست شناسان و به خصوص تکامل شناسان به شمار مي رود؛ به عبارت ديگر، گرچه قيافه ي حق به جانب تکامل شناسان و ادعاي آن ها در مورد آناليزهاي ژنتيکي شان، ممکن است مخاطبان (به خصوص مخاطبان عام) را فريب دهد، اما اين مسئله، تضادها، بحث ها و چالش هاي جدي در بين دانشمندان به جريان انداخته است که هنوز تا رفع آن ها، فاصله ي زيادي وجود دارد!
البته « تکامل شناسان » که دروغ، دغل و فريبکاري بخش بزرگي از حياتشان را تشکيل مي دهد، معمولاً با ارايه ي درخت هاي فيلوژنتيک هدف دار، سعي مي کنند تا ساير درخت هاي فيلوژنتيک را که با ادعاهايشان تناقض دارد، سانسور نموده و به لطايف الحيل، از چشم مخاطبان خود دور بدارند! در واقع آن ها در مقالات خود که در دفاع از « فرضيه ي تکامل » مي نگارند، فقط به ارايه ي درخت هاي فيلوژنتيکي مبادرت مي ورزند که نشان مي دهد ادعاهاي آن ها درست مي باشد! و درخت هاي فيلوژنتيک متناقض يا ناهماهنگ با ادعاهايشان را سانسور مي نمايند! براي درک بهتر اين مسئله، مجدداً مثال مربوط به مقايسه ي ژن هاي « پلاسموديوم ها » را از اين زاويه نيز بررسي مي نماييم:
فرض مي کنيم که تکامل شناسان بخواهند اثبات نمايند که انگل هاي دو گونه ي « پلاسموديوم الونگاتوم : P. elongatum » و « پلاسموديوم گاليناسئوم : P. gallinaceum »، از نظر تکاملي و ژنتيکي به يکديگر نزديک هستند! آن ها براي اثبات اين ادعاي خودشان، در مقالات مربوط به فرضيه ي تکامل، فقط آناليز به دست آمده از « DNA ميتوکندريال (mtDNA) کد کننده ي پروتئين سيتوکروم b موسوم به (Cytochrome b) » را که مطابق ادعاهايشان بوده و شباهت ژنتيکي دو گونه ي « پلاسموديوم الونگاتوم : P. elongatum » و « پلاسموديوم گاليناسئوم : P. gallinaceum » را به نمايش مي گذارد، به مخاطبان خود عرضه مي کنند!
به عبارت ديگر، آن ها با زيرکي تمام، تنها درخت فيلوژنتيک به دست آمده از « DNA ميتوکندريال (mtDNA) کد کننده ي پروتئين سيتوکروم b موسوم به (Cytochrome b) » را که مطابق ادعاهايشان است، ارايه مي کنند؛ حال آن که درخت هاي فيلوژنتيک ناشي از آناليز بخش هاي ديگر DNA و ژن هاي ديگر « پلاسموديوم ها » را که با ادعاهايشان تناقض و ناهماهنگي دارد، پنهان مي کنند! بدين ترتيب که آناليز « DNA پلاستيدي کد کننده ي پروتئاز کازئينوليتيک موسوم به (ClpC) » را که با ادعاهايشان ناهماهنگ بوده و دو گونه ي « پلاسموديوم الونگاتوم : P. elongatum » و « پلاسموديوم گاليناسئوم : P. gallinaceum » را به نسبت دور از هم نشان مي دهد، در کتب و مقالاتشان به نمايش نمي گذارند!:
فرض مي کنيم که تکامل شناسان بخواهند اثبات نمايند که انگل هاي دو گونه ي « پلاسموديوم الونگاتوم : P. elongatum » و « پلاسموديوم گاليناسئوم : P. gallinaceum »، از نظر تکاملي و ژنتيکي به يکديگر نزديک هستند! آن ها براي اثبات اين ادعاي خودشان، در مقالات مربوط به فرضيه ي تکامل، فقط آناليز به دست آمده از « DNA ميتوکندريال (mtDNA) کد کننده ي پروتئين سيتوکروم b موسوم به (Cytochrome b) » (درخت وسط) را که مطابق ادعاهايشان بوده و شباهت ژنتيکي دو گونه ي « پلاسموديوم الونگاتوم : P. elongatum » و « پلاسموديوم گاليناسئوم : P. gallinaceum » را به نمايش مي گذارد، به مخاطبان خود عرضه مي کنند! به عبارت ديگر، آن ها با زيرکي تمام، تنها درخت فيلوژنتيک به دست آمده از « DNA ميتوکندريال (mtDNA) کد کننده ي پروتئين سيتوکروم b موسوم به (Cytochrome b) » را که مطابق ادعاهايشان است، ارايه مي کنند (درخت وسط)؛ حال آن که درخت هاي فيلوژنتيک ناشي از آناليز بخش هاي ديگر DNA و ژن هاي ديگر « پلاسموديوم ها » را که با ادعاهايشان تناقض و ناهماهنگي دارد، پنهان مي کنند! بدين ترتيب که آناليز « DNA پلاستيدي کد کننده ي پروتئاز کازئينوليتيک موسوم به (ClpC) » را که با ادعاهايشان ناهماهنگ بوده و دو گونه ي « پلاسموديوم الونگاتوم : P. elongatum » و « پلاسموديوم گاليناسئوم : P. gallinaceum » را به نسبت دور از هم نشان مي دهد (درخت سمت چپ)، در کتب و مقالاتشان مخفي مي نمايند!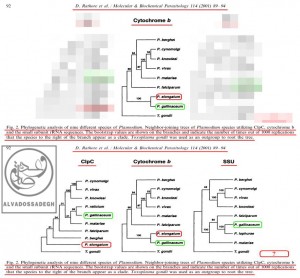
در مثالي ديگر، فرض مي کنيم که تکامل شناسان تلاش مي کنند تا به مخاطبان خود اثبات نمايند که انگل هاي دو گونه « پلاسموديوم گاليناسئوم : P. gallinaceum » و « پلاسموديوم مالاريه : P. malariae »، از نظر تکاملي و ژنتيکي به يکديگر نزديک هستند!
آن ها براي اثبات اين ادعاي خودشان، در مقالات مربوط به فرضيه ي تکامل، فقط آناليز به دست آمده از « DNA پلاستيدي کد کننده ي پروتئاز کازئينوليتيک موسوم به (ClpC) » را که مطابق ادعاهايشان بوده و شباهت ژنتيکي دو گونه ي « پلاسموديوم گاليناسئوم : P. gallinaceum » و « پلاسموديوم مالاريه : P. malariae » را به نمايش مي گذارد، به مخاطبان خود عرضه مي کنند!
به عبارت ديگر، آن ها با زيرکي تمام، تنها درخت فيلوژنتيک به دست آمده از « DNA پلاستيدي کد کننده ي پروتئاز کازئينوليتيک موسوم به (ClpC) » را که مطابق ادعاهايشان است، ارايه مي کنند؛ حال آن که درخت هاي فيلوژنتيک ناشي از آناليز بخش هاي ديگر DNA و ژن هاي ديگر « پلاسموديوم ها » را که با ادعاهايشان تناقض و ناهماهنگي دارد، پنهان مي نمايند!
بدين ترتيب که آناليز « DNA ميتوکندريال (mtDNA) کد کننده ي پروتئين سيتوکروم b موسوم به (Cytochrome b) » و نيز آناليز « DNA هسته اي کد کننده ي زير واحد کوچک RNA ريبوزومي (rRNA) موسوم به (SSU) » را که با ادعاهايشان ناهماهنگ بوده و دو گونه ي « پلاسموديوم گاليناسئوم : P. gallinaceum » و « پلاسموديوم مالاريه : P. malariae » را به نسبت دور از هم نشان مي دهد، در کتب و مقالاتشان به نمايش نمي گذارند و از ديد اکثر مخاطبان (به خصوص مخاطبان عام)، مخفي مي نمايند!:
فرض مي کنيم که تکامل شناسان بخواهند اثبات نمايند که انگل هاي دو گونه « پلاسموديوم گاليناسئوم : P. gallinaceum » و « پلاسموديوم مالاريه : P. malariae »، از نظر تکاملي و ژنتيکي به يکديگر نزديک هستند! آن ها براي اثبات اين ادعاي خودشان، در مقالات مربوط به فرضيه ي تکامل، فقط آناليز به دست آمده از « DNA پلاستيدي کد کننده ي پروتئاز کازئينوليتيک موسوم به (ClpC) » (درخت سمت چپ) را که مطابق ادعاهايشان بوده و شباهت ژنتيکي دو گونه ي « پلاسموديوم گاليناسئوم : P. gallinaceum » و « پلاسموديوم مالاريه : P. malariae » را به نمايش مي گذارد، به مخاطبان خود عرضه مي کنند! به عبارت ديگر، آن ها با زيرکي تمام، تنها درخت فيلوژنتيک به دست آمده از « DNA پلاستيدي کد کننده ي پروتئاز کازئينوليتيک موسوم به (ClpC) » را که مطابق ادعاهايشان است، ارايه مي کنند (درخت سمت چپ)؛ حال آن که درخت هاي فيلوژنتيک منبعث از آناليز بخش هاي ديگر DNA و ژن هاي ديگر « پلاسموديوم ها » را که با ادعاهايشان تناقض و ناهماهنگي دارد، پنهان مي نمايند! بدين ترتيب که آناليز « DNA ميتوکندريال (mtDNA) کد کننده ي پروتئين سيتوکروم b موسوم به (Cytochrome b) » (درخت وسط) و نيز آناليز « DNA هسته اي کد کننده ي زير واحد کوچک RNA ريبوزومي (rRNA) موسوم به (SSU) » (درخت سمت راست) را که با ادعاهايشان ناهماهنگ بوده و دو گونه ي « پلاسموديوم گاليناسئوم : P. gallinaceum » و « پلاسموديوم مالاريه : P. malariae » را به نسبت دور از هم نشان مي دهد، در کتب و مقالاتشان به نمايش نمي گذارند!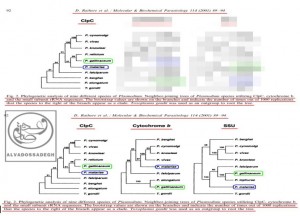
در واقع تکامل شناسان همچون ماهي لغزنده اي که از دست ماهيگير فرار مي کند و از دست صياد مي گريزد، تلاش مي کنند تا با چنين حربه هايي از دست منتقدان احتمالي بگريزند و شواهد علمي که ممکن است از سوي منتقدان عليه آن ها استفاده شود را تا حد امکان در بايکوت رسانه اي قرار دهند! ان شاء الله در سلسله مقالات آتي، مثال هاي عملي و عيني چنين فريبکاري هايي را خدمت مخاطبان محترم، ارايه خواهيم نمود!
البته چنين تاکتيک هايي ديگر سودي براي آن ها نخواهد داشت! چرا که با رشد علمي جوامع و نيز دسترسي بيشتر دانشمندان به منابع مختلف، پنهان کاري هاي علمي، بسيار سخت تر از قبل خواهد بود!
با توجه به مطالبي که تاکنون بيان شد، مي توان دريافت که گرچه مطالعه ي فسيل ها با استفاده از آناليز ژني مبتني بر پروتکل « DNA باستاني : Ancient DNA »، دقيق تر از مطالعه بر اساس آناتومي و ظاهر فسيل ها مي باشد، اما اين روش نيز علي رغم ظاهر جذاب، فريبنده و دهان پرکني که دارد، با ايرادات، اشکالات و ابهامات بي شماري مواجه است که اعتماد به آن را نيز غير ممکن مي کند!
ما در صفحات قبل، موارد متعددي از ضعف ها و ايرادات مربوط به مطالعه ي فسيل ها بر مبناي روش « DNA باستاني : Ancient DNA » را برشمرديم و مطالعات بيشتر در اين زمينه را به مخاطبان محترم واگذار مي کنيم. البته اين ضعف ها و اشکالات تنها توهم! ما نيست و محققان ديگر نيز به اشکالات اين روش، اشاره کرده اند و حتي دانشمندان و دانش پژوهان را به دقت بيشتر در اين گونه اشکالات، حفظ روحيه ي انتقادي در مورد اين گونه مطالعات و نيز کنکاش جدي در مورد باورپذيري، صحت و قابل اطمينان بودن نتايج حاصل از اين تحقيقات، دعوت نموده اند!:
بسياري از محققان، به اشکالات موجود در مطالعه ي فسيل ها به روش « DNA باستاني : Ancient DNA » ، اشاره کرده اند و حتي دانشمندان و دانش پژوهان را به دقت بيشتر در اين گونه اشکالات، حفظ روحيه ي انتقادي در مورد اين گونه مطالعات و نيز کنکاش جدي در مورد باورپذيري، صحت و قابل اطمينان بودن نتايج حاصل از اين تحقيقات، دعوت نموده اند!
اما علي رغم مسايلي که گفته شد، مشکل بزرگي دامن گير جامعه ي علمي کشورمان شده است که همانا عدم وجود اعتماد به نفس و عدم پرورش روحيه ي انتقادي در بسياري از دانش پژوهان و دانشمندان اين مرز و بوم مي باشد! اين امر به خصوص در مواردي که اصطلاحات جذاب و فريبنده اي همچون « ژنتيک »، « DNA »، « پروتئوميکس »، « بيوتکنولوژي »، « نانوتکنولوژي »، « سلول هاي بنيادي » و … از سوي دانشمندان غربي عنوان مي شود، بيش از پيش خود را نشان مي دهد! به نحوي که جامعه ي آکادميک کشورمان که سرشار از دانشجويان و اساتيد با هوش و با سواد نيز مي باشد، در مقابل اين اصطلاحات و ادعاهاي دانشمندان غربي در اين زمينه ها، تسليم شده و بسياري از ادعاهاي آن ها را چشم بسته و بدون تفکر مي پذيرد!
البته اين ايراد بيمارگونه مختص جامعه ي علمي ما نيست! بلکه مشابه همين روند، با شدتي کمتر در جوامع غربي نيز مشاهده مي گردد! به نحوي که برخي از دانشمندان متفکر و دغدغه مند نيز زبان به اعتراض در اين زمينه گشوده و جامعه ي علمي را از اعتماد بي محابا و بدون تفکر نسبت به متدهاي جديد « مهندسي ژنتيک » و مطالعات مبتني بر چنين روش هايي، بر حذر داشته و خواهان حاکميت مجدد تفکر بر جوامع علمي شده اند!
براي مثال، در مقاله ي زير که در نقد پروژه ي بين المللي معروف ژنتيکي موسوم به « HapMap » نگاشته شده و در سال 2006 ميلادي در نشريه ي معروف « مجله ي اروپايي ژنتيک انساني : European Journal of Human Genetics » وابسته به انتشارات مشهور « Nature Publishing Group » منتشر شده است، مولفان مقاله، به صراحت، ادعاها و سر و صداهاي زيادي که در مورد پروژه ي ژنتيکي بين المللي « HapMap » به راه افتاده است را زير سوال برده و از حمله ي فله اي! و گروهي! دانشمندان به سمت « مطالعات ژنتيکي »، آن هم بدون اين که پشتوانه ي فکري دقيق و عالمانه اي، زمينه ساز بسياري از اين مطالعات باشد، به شدت ابراز ناراحتي و گلايه نموده اند! به نحوي که در بخش انتهايي اين مقاله، چنين مطلبي را درج نموده اند:(207)
»It would be far better to spend more time thinking and planning before jumping in to genotyping every sample we can get our hands on. «
« بسيار بهتر خواهد بود که ما وقت بيشتري را به تفکر و برنامه ريزي اختصاص دهيم، قبل از اين که هر نمونه اي که به دستمان برسد را فوراً آناليز ژنتيکي نماييم! »(207)
دانشمندان متفکر و دغدغه مند نيز، زبان به اعتراض گشوده و جامعه ي علمي را از اعتماد بي محابا و بدون تفکر نسبت به متدهاي جديد « مهندسي ژنتيک » و مطالعات مبتني بر چنين روش هايي، بر حذر داشته و خواهان حاکميت مجدد تفکر بر جوامع علمي شده اند! براي مثال، در مقاله ي فوق که در نقد پروژه ي بين المللي معروف ژنتيکي موسوم به « HapMap » نگاشته شده و در سال 2006 ميلادي در نشريه ي معروف « مجله ي اروپايي ژنتيک انساني : European Journal of Human Genetics » وابسته به انتشارات مشهور « Nature Publishing Group » منتشر شده است، مولفان مقاله، به صراحت، ادعاها و سر و صداهاي زيادي که در مورد پروژه ي ژنتيکي بين المللي « HapMap » به راه افتاده است را زير سوال برده و از حمله ي فله اي! و گروهي! دانشمندان به سمت « مطالعات ژنتيکي »، آن هم بدون اين که پشتوانه ي فکري دقيق و عالمانه اي، زمينه ساز بسياري از اين مطالعات باشد، به شدت ابراز ناراحتي و گلايه نموده اند! به نحوي که در بخش انتهايي اين مقاله، چنين مطلبي را درج نموده اند: « بسيار بهتر خواهد بود که ما وقت بيشتري را به تفکر و برنامه ريزي اختصاص دهيم، قبل از اين که هر نمونه اي که به دستمان برسد را فوراً آناليز ژنتيکي نماييم! »
چنين انتقاداتي آن هم نسبت به پروزه هاي مشهوري هم چون « HapMap » که ارتباط مستقيمي هم با مقوله ي « تکامل » ندارد و بيشتر در حيطه ي ژنتيک پزشکي مطرح مي گردد و تعريف ها و تمجيدهاي بي شمار کورکورانه اي از سراسر جهان (حتي از کشور ما!) را نيز نسبت به خود بر مي انگيزد، يک درس بسيار خوب براي ما دارد: « بسياري از پروژه هاي مشهور علمي، متد پيشرفته، اما تفکر و برنامه ي ضعيفي دارند! و متاسفانه اين مطالعات، با اصطلاحات دهان پرکن و روش هاي جديدشان، تلاش مي کنند تا عقبه ي ضعيف فکري و خلاء برنامه ريزي دقيق خود را بپوشانند! ».
ما دانشمندان و دانش پژوهان ايران زمين، نبايد تنها نظاره گر باشيم! هنگامي که اصطلاحات فريبنده و جذابي چون « مطالعه ي ژنتيک »، مطالعه بر روي « DNA »، مطالعه به روش « DNA باستاني : Ancient DNA » و … را مي شنويم، نبايد تصور کنيم که استفاده از اين روش ها در مطالعات، به معناي دقت و صحت نتايج اين مطالعات است! همچنين نبايد تصور کنيم که ادعاي محققان مذکور درباره ي نتايج مطالعات، واقعاً و حقيقتاً با آن چه که مطالعه در عمل ارايه مي کند، مترادف است! چه بسا مطالعات متعدد ژنتيکي با روش هاي پيشرفته انجام مي شوند، اما به دليل ضعف عقبه ي فکري، نتايجي که به دست مي دهند، ارزشي ندارد!
به هر حال با توجه به مطالبي که گفته شد، نتيجه مي گيريم که مطالعه ي فسيل ها به روش « DNA باستاني : Ancient DNA »، گرچه از روش مطالعه ي ظاهري فسيل ها، بهتر و منطقي تر مي باشد، ولي اين روش نيز با مشکلات، چالش ها، ابهامات و ضعف هاي متعددي رو به رو است و به نتايج حاصل از آن نيز نمي توان اعتماد کرد! بنابراين به اين روش بايد به مثابه کودک نوزادي نگاه کرد که راهي بسيار طولاني تا بالغ شدن و قابل اعتماد گرديدن در پيش دارد!
ادامه دارد …
خادم الامام (عج) – وعده صادق
بخش بعد: آيا ادعاهاي موجود پيرامون اطلاعات کسب شده ازنئاندرتا ها صحیح است؟… DNA
منابع و مآخذ
Green, R. E., et al. (2008). A complete Neandertal mitochondrial genome sequence determined by high-throughput sequencing. Cell, 134, 416–426.
Krings, M; Stone, A; Schmitz, R W; Krainitzki, H; Stoneking, M; Paabo, S. Neandertal DNA sequences and the origin of modern humans. Cell 90 (1997): 19-30.
Noonan JP, Coop G, Kudaravalli S, Smith D, Krause J, et al. (2006) Sequencing and analysis of Neanderthal genomic DNA. Science 314: 1113-1118.
Green RE, Krause J, Ptak SE, Briggs AW, Ronan MT, et al. (2006) Analysis of one million base pairs of Neanderthal DNA. Nature 16: 330-336.
Ankel-Simons F, Cummins JM. Misconceptions about mitochondria and mammalian fertilization: implications for theories on human evolution. ProcNatlAcadSci U S A. 1996 Nov 26; 93(24):13859-63.
Schwartz M, Vissing J (2002). Paternal inheritance of mitochondrial DNA. N Engl J Med 347: 576–580.
Gibbons A (January 1998), “Calibrating the mitochondrial clock”, Science 279 (5347): 28–9.
http://www.dnai.org/teacherguide/pdf/reference_romanovs.pdf
Tully, L.A. et al., A sensitive denaturing gradient-gel electrophoresis assay reveals a high frequency of heteroplasmy in hypervariable region 1 of the human mitochondrial DNA control region. Am. J. Hum. Genet., 2000; 67: 432-443.
Calloway CD, Reynolds RL, Herrin GL, Anderson WW: The frequency of heteroplasmy in the HVII region of mtDNA differs across tissue types and increases with age. Am J Hum Genet 2000, 66(4):1384-1397.
http://ocw.tufts.edu/Content/20/CourseHome/301011/301031
http://www.newcastle-mitochondria.com/mitochondria/what-do-mitochondria-do/
Calloway CD, Reynolds RL, Herrin GL, Anderson WW: The frequency of heteroplasmy in the HVII region of mtDNA differs across tissue types and increases with age. Am J Hum Genet 2000, 66(4):1384-1397.
Sondheimer N, Glatz CE, Tirone JE, Deardorff MA, Krieger AM, Hakonarson H: Neutral mitochondrial heteroplasmy and the influence of aging. Hum Mol Genet 2011, 20(8):1653-1659.
https://www.promega.com/~/media/files/resources/conference%20proceedings/ishi%2010/oral%20presentations/35calloway.pdf?la=en
Gibbons A (January 1998), “Calibrating the mitochondrial clock”, Science 279 (5347): 28–9.
http://www.dnai.org/teacherguide/pdf/reference_romanovs.pdf
Howell N, Howell C, Elson JL (2008) Time dependency of molecular rate estimates for mtDNA: this is not the time for wishful thinking. Heredity, 101, 107–108.
Gibbons A (January 1998), “Calibrating the mitochondrial clock”, Science 279 (5347): 28–9.
http://www.dnai.org/teacherguide/pdf/reference_romanovs.pdf
Howell N, Howell C, Elson JL (2008) Time dependency of molecular rate estimates for mtDNA: this is not the time for wishful thinking. Heredity, 101, 107–108.
White DJ, Wolff JN, Pierson M and Gemmell NJ (2008) Revealing the hidden complexities of mtDNA inheritance. Molecular Ecology 17: 4925–4942.
http://epidemic.bio.ed.ac.uk/how_to_read_a_phylogeny
http://guava.physics.uiuc.edu/~nigel/courses/598BIO/498BIOonline-essays/hw2/files/hw2_li.pdf
http://www.answers.com/topic/phylogenetic-tree
http://en.wikipedia.org/wiki/Phylogenetic_tree
Pearson WR, Robins G, Zhang T. Generalized neighbor-joining: more reliable phylogenetic tree reconstruction. MolBiolEvol. 1999 Jun;16(6):806-16.
http://www.icp.ucl.ac.be/~opperd/private/neighbor.html
http://www.answers.com/topic/neighbor-joining
http://en.wikipedia.org/wiki/Neighbor_joining
http://stat.duke.edu/people/theses/WuY.html
http://www.answers.com/topic/bayesian-inference-in-phylogeny
http://en.wikipedia.org/wiki/Bayesian_inference_in_phylogeny
Rathore, D., A. M. Wahl, M. Sullivan, and T. F. McCutchan. 2001. A phylogenetic comparison of gene trees constructed from plastid mitochondrial and genomic DNA of Plasmodium species. Mol. Biochem. Parasitol. 114:89-94.
Jeffrey R, Eric CR (2007) Review of Phylogenetic Tree Construction University
of Louisville Bioinformatics Laboratory Technical Report Series 1-7.
Bapteste, E., E. Susko, J. Leigh, D. MacLeod, et al. 2005. “Do orthologous gene phylogenies really support tree-thinking?” Bmc Evolutionary Biology 5: 33-33.
Gilbert, T., Bandelt H.-J., Hofreiter, M. and Barnes I. (2005) Assessing ancient DNA studies. Trends in Ecology and Evolution, 20, 541-544.
Terwilliger JD, Hiekkalinna T (2006) An utter refutation of the “fundamental theorem of the HapMap”. Eur J Hum Genet 14: 426-437.
منبع : http://www.alvadossadegh.com/fa/vijhe-13/17113-217113.html
 آرمان مهدویت مذهبی ، اعتقادی
آرمان مهدویت مذهبی ، اعتقادی


